Con il termine plasmide vengono definite delle molecole circolari di DNA presenti nei batteri. Queste molecole sono dotate di un’origine per la replicazione e sono molto simili a dei cromosomi batterici, ma sono più piccole e non sono strettamente necessarie alla vita del batterio. I plasmidi portano però dei geni in singola copia, che rappresentano un corredo genetico aggiuntivo per il batterio che ne possiede, e possono quindi fornirgli delle caratteristiche specifiche. Dei geni comunemente contenuti nei plasmidi sono quelli che conferiscono al batterio la resistenza agli antibiotici.
I plasmidi possono quindi essere replicati all’interno dal batterio, e i loro geni espressi, indipendentemente dal cromosoma principale. Essi vengono trasmessi alle cellule figlie nella divisione cellulare, conferendo lo stesso vantaggio a tutte le cellule figlie.
Tutte queste caratteristiche, unite alla conoscenza di alcuni enzimi batterici (enzimi di restrizione) in grado di tagliare il DNA in corrispondenza di sequenze specifiche, hanno portato i biologi molecolari ad utilizzare i plasmidi per introdurre nei batteri del DNA a loro estraneo, al fine di produrre proteine oppure per amplificare tratti di DNA.
Inserendo un gene per una proteina in un plasmide, ed immettendo il plasmide in un batterio, posso conferire a tutte le cellule figlie di quel batterio la capacità di sintetizzare la proteina, della quale posso quindi produrre quantità a piacere.
Qui vediamo uno schema dei processi di clonaggio di un plasmide in una cellula batterica.
A partire dagli anni settanta vennero prodotti in fabbrica plasmidi sulla base di quelli esistenti in natura, aggiungendo frammenti di DNA presi da altri organismi, batteri e non. Successivamente il livello di “ingegnerizzazione” si è sofisticato sempre di più, tanto che oggi si trovano in commercio plasmidi del tutto artificiali. Oltre ai plasmidi batterici (come pBR322) vengono utilizzati per costruire plasmidi anche cromosomi di virus che normalmente infettano i batteri (per esempio Lambda o M13), perché essi sono in grado di inserirsi nel cromosoma batterico e permettono di amplificare frammenti di DNA estraneo molto lunghi (fino a cinquantamila paia di basi). Il termine plasmide si è quindi esteso a tutte quelle molecole usate come “trasportatori” di DNA, che si definiscono quindi anche vettori.
I frammenti di DNA aggiunti in fabbrica sono necessari ad espletare specifiche funzioni, e devono essere disposti in un preciso ordine affinché le funzioni vengano espletate correttamente.
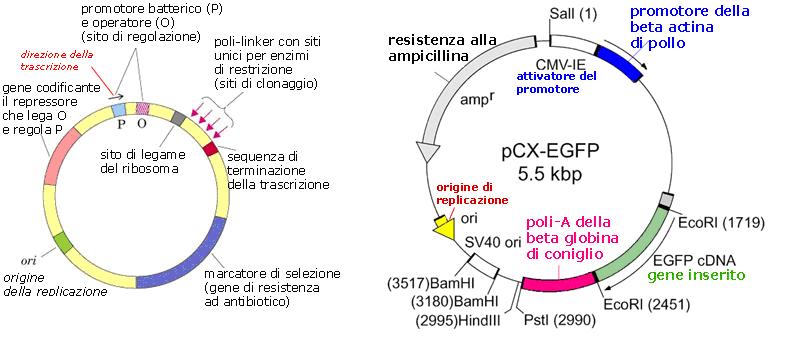
Qui di seguito vediamo uno schema generico di mappa di vettore di espressione genica, e la mappa di un vettore vero, il pCX-EGFPTM, lungo 5,5 kilobasi (kbp), a titolo di esempio. I numeri tra parentesi indicano il numero della base di inizio del gene o sito di restrizione indicato. EcoRI, PstI, BamHI, SalI sono sigle che indicano i siti per gli enzimi di restrizione. Naturalmente le varianti allo schema fondamentale sono moltissime.
Innanzitutto vengono messi dei geni per la resistenza a vari antibiotici, che permettono di selezionare solo quei batteri che hanno recepito il plasmide. I batteri trasformati con il plasmide contenente DNA estraneo vengono fatti crescere su terreni contenenti uno o più antibiotici, e così solo quelli che hanno il plasmide possono sopravvivere, mentre gli altri muoiono. I geni per la resistenza agli antibiotici sono detti marcatori di selezione.
Altri frammenti aggiunti in fabbrica contengono delle sequenze che permettono il taglio del plasmide usando gli enzimi di restrizione. Il ricercatore può così aprire il plasmide ed inserirvi il gene estraneo, a sua volta ottenuto grazie ad un taglio con lo stesso enzima di restrizione. In uno stesso plasmide vi sono siti per diversi enzimi di restrizione, per dare più scelta allo sperimentatore, ma sempre in un’unica copia, per evitare che il plasmide venga tagliuzzato; i siti si trovano di solito concentrati tutti in una stessa zona del plasmide. Vi possono anche essere alcuni altri siti per enzimi di restrizione diversi da quelli per l’inserzione, e disposti in due zone del plasmide, da usare nel caso si volesse rimuovere il gene inserito.
Vi sono poi segnali necessari ad avviare della sintesi di RNA messaggero (mRNA), cioè la trascrizione, a partire dal gene estraneo, e segnali di avvio della traduzione di mRNA in catena polipeptidica. Tutti questi segnali, che sono sempre delle sequenze specifiche di nucleotidi, devono trovarsi a monte del gene da esprimere, quindi prima dell’estremità 5’ del sito di inserzione del gene.
La sequenza che serve da avvio della trascrizione si chiama promotore. Esso è una specifica sequenza di DNA che viene riconosciuta dall’enzima RNA polimerasi, e può derivare o da batteri, cioè da procarioti, se il DNA da utilizzare per la produzione di proteine è procariotico, oppure da eucarioti se i geni per le proteine da produrre provengono da organismi superiori diversi dai batteri. Dato l’elevato numero di studi condotti sull’uomo e sul topo, molti plasmidi hanno promotori provenienti da queste due specie, e per l’espressione dei geni vengono utilizzate cellule eucariotiche, per esempio lieviti, cellule di scimmia, di topo, di uomo. Ciò si rende necessario in quanto la proteina eucariotica necessita della maturazione del mRNA, e di modificazioni post-traduzionali come l’aggiunta di catene di zuccheri o altri gruppi chimici, o la creazione di ponti disolfuro tra amminoacidi, tutti processi che non possono avvenire nella cellula procariotica.
Vi saranno poi dei segnali di stop dopo l’estremità 3’ del gene inserito: prima lo stop della traduzione di mRNA in proteina, e poi lo stop della trascrizione in RNA, il terminatore, in modo da consentire la trascrizione di tutto il gene. Il terminatore è costituito da una breve sequenza palindromica posta 15-20 basi prima della fine del trascritto, e da una coda di poli-A.
La maturazione dell’RNA, o splicing, serve ad eliminare le sequenze del gene eucariotico non codificanti (introni) e a cucire tra loro quelle codificanti (esoni). Una volta maturo, l’RNA può essere tradotto in proteina.
Le varianti allo schema di base sono moltissime, a seconda delle esigenze di studio.
Nel plasmide possono essere inserite delle piccole code codificanti per amminoacidi particolari (per esempio istidina), sia all’estremità 5’ che 3’ del prodotto trascritto. La presenza di code di amminoacidi nella proteina ne permette la efficiente purificazione con metodi specifici.
I vettori di ultima generazione sono ottimizzati anche per una più efficiente lettura del codice nel giusto frame, così da evitare la produzione di proteine anomale dovuto semplicemente al un errore di lettura del codice genetico. Alcuni portano anche un gene per la luciferasi, una proteina fluorescente, derivante da batteri o insetti, che viene trascritta e sintetizzata insieme a quella di interesse; essa serve a dimostrare l’avvenuto giusto funzionamento del sistema di espressione. Ciò permette una verifica immediata dell’espressione, direttamente sulle piastre di coltura delle cellule, in quegli esperimenti dove sia di grande importanza una risposta di tipo positivo o negativo, senza bisogno di fare complesse analisi mirate per verificare la presenza della proteina di interesse. Questi sistemi sono usati anche per studiare i promotori, o gli operatori che regolano l’espressione genica.
Libri
Biologia Molecolare del Gene. J.D. Watson et al. Ed. Zanichelli.
Ingegneria Genetica E. Boncinelli, A. Simeone, P. Iaccarino Idelson. Ed. Idelson-Gnocchi.
Le biotecnologie. M. Buiatti, Ed. Il Mulino, Collana “Farsi un’idea”, n.65.
Per la distinzione tra procarioti ed eucarioti:
http://www.vialattea.net/esperti/php/risposta.php?num=3384
http://www.vialattea.net/esperti/bio/endosimbiosi/
Cercare o amplificare un gene; organismi geneticamente modificati
http://www.vialattea.net/esperti/php/risposta.php?num=6355
http://www.vialattea.net/esperti/php/risposta.php?num=4304
Plasmidi, vettori, ingegneria genetica
http://biotech-adventure.okstate.edu/low/handson/cloning/
http://www.molecularlab.it/post-genomica/proteomica/index.asp
http://www.molecularlab.it/clonaggio/vettori/index.asp